BDMLファイルに記録された細胞核をブラウザで見られるようにする
生命現象を記録し、データベースを構築するというプロジェクトが理研で行われている。理研の広報ページが詳しい。私は知らなかったが、説明を読むとなにやらすごそうなプロジェクトに聞こえる。
ところでこのBDMLファイル、このような感じで誰でもダウンロードできるように整備されているのだが、これをWebブラウザで可視化したいという要望があった。聞くところによると、こちらのファイルにはXML形式で核の輪郭が格納されているらしい。しばらく前にバイナリ形式のシミュレーションデータが格納された謎のファイルを可視化するという仕事を受注したことがあり、そのときは仕様書が渡されなかったのでバイナリエディタを片手に謎解き気分で取り組んだ。しかし今度は仕様がわりとはっきりしているし、中身は巨大なXMLなので少なくとも読むことはできる。とはいえ普通のテキストエディタで開いてみてもクラッシュしてしまうくらいの大きさではある。となるとXMLをそのままブラウザでダウンロードし、それをパースして3DCGに変換するのはなかなかヘヴィな処理になる。100MB以上あるXMLのパースをブラウザ内で毎度毎度やるのはつらそうだ。
そんなわけで、とりあえずBDMLから不要なデータを削除しないといけない。NodeJsのオブジェクトとして変換したBDMLファイルは以下のような中身を持っている。
{ version: '0.18',
xmlns: 'http://ssbd.qbic.riken.jp/bdml',
'xmlns:xsi': 'http://www.w3.org/2001/XMLSchema-instance',
'xsi:schemaLocation': 'http://ssbd.qbic.riken.jp/bdml http://ssbd.qbic.riken.jp/bdml/bdml0.18.xsd',
info:
[ { bdmlID: [Array],
title: [Array],
version: [Array],
release: [Array],
license: [Array] } ],
summary:
[ { description: [Array],
datatype: [Array],
organism: [Array],
localID: [Array],
basedon: [Array],
contributors: [Array],
PMID: [Array],
dblink: [Array] } ],
contact:
[ { name: [Array],
'E-mail': [Array],
organization: [Array],
department: [Array],
laboratory: [Array],
address: [Array] } ],
methods: [ { summary: [Array], source: [Array], pdpml: [Array] } ],
data: [ { scaleUnit: [Array], object: [Array], component: [Array] } ] }
このうち、描画に必要なデータはdata[0].componentに入っており、その中はさらに以下のような配列になっている。
[ { componentID: [ '1000' ],
componentName: [ 'P0' ],
time: [ '1' ],
measurement: [ [Object] ] },
{ componentID: [ '2000' ],
componentName: [ 'P0' ],
time: [ '2' ],
prevID: [ '1000' ],
measurement: [ [Object] ] },
{ componentID: [ '3000' ],
componentName: [ 'P0' ],
time: [ '3' ],
prevID: [ '2000' ],
measurement: [ [Object] ] },
{ componentID: [ '4000' ],
componentName: [ 'P0' ],
time: [ '4' ],
prevID: [ '3000' ],
measurement: [ [Object] ] },
....
このように中身を確認しながらどんどん掘り進めていき、やっとのことでxyz座標が手に入る。
// docs.data[0].component[0].measurement[0].line[0].xyzSequence[0].xyz
[ { x: [ '302' ], y: [ '261' ], z: [ '19' ] },
{ x: [ '301' ], y: [ '262' ], z: [ '19' ] },
{ x: [ '301' ], y: [ '263' ], z: [ '19' ] },
{ x: [ '301' ], y: [ '264' ], z: [ '19' ] },
{ x: [ '301' ], y: [ '265' ], z: [ '19' ] },
...
このように非常に深いネストになっていたものを整形し、輪郭を構成する点群の座標だけ取り出す。JSON形式に変換したのでたくさん容量を取る閉じタグがなくなり、Stringで格納されていた数値もIntに変換する。結果的に容量は半分以下に削減できた。NodeJsで全ての処理を行ったが、XMLのパースを行うライブラリの大半は大きすぎるXMLデータの読み込みに対応していない。唯一pixl-xmlだけが危なげなく高速に読み込めた。他のライブラリに比べスター数もダウンロード数も少ないが、これが一番よさそうだった。メモリ消費量は少なく読み取りは速い。文句の付けようがない。こんなものを無料で公開してしまっていいのだろうか。デフレ社会である。
このBDMLファイルには複数のタイムステップと細胞系譜における名付け(AB、ABa、ABp、P1、P2、EMS…)が含まれているが、それらを別個に表示させられるように細胞の名前でgroupByし、ファイルを分ける。そしてそのファイルの中でシンプルな配列を作成し、それをそのまま時系列として使えるようにした。
さて、輪郭の座標群が細胞系譜とタイムステップ別にまとめられたファイルが生成できた。次はこれを3D空間に描画して見られるようにする必要がある。いまあるファイルをそのまま表示しても、輪郭だけが表示されるので細胞核の形状はよくわからない。とはいえなんとなく丸い形状は確認できるので、ちゃんと読み取れているのだろう。この輪郭だけの情報からポリゴンを作り出す必要がある。実のところポリゴンデータを作り出す方法としてメジャーな手法は限られており、当たり前のようにマーチングキューブ法を使う。なんとなく自明な気もするアルゴリズムだが、実際にスクラッチから作るのはかなり難しい。しかしすでに多数の実装があるので既存のコードやライブラリをありがたく使わせていただこう。
つぎの問題は、元の輪郭データには輪郭しかふくまれていないことだ。輪郭だけをポリゴンデータにしても、それは複数の輪っかが生成されるだけで意味はない。つまり輪郭の中身を埋めてあげる必要がある。こちらはそれほど難しい話ではなく、輪郭データなのだから単純に内側をペイントツールのように塗りつぶしてあげればよい。輪郭の内側を探すのが面倒であれば、最初から1で埋めた2次元配列に輪郭座標の場所を0でうめ、そしてその周りも0で埋めれば簡単に完成する。座標[0, 0, 0]に輪郭が位置しないということは簡単に調べられるので、塗りつぶしの開始地点は原点でよいのだろう。
ここまでくるとだいぶ細胞の形は見えてくる。あとはできあがったポリゴンデータに色を付けたりカメラ位置を変更できるようにしたりして使いやすくしていく。ThreeJsは恐ろしく巨大なライブラリなので、基本的にCDNからロードしたほうがいいだろう。OrbitControlsも同様にやる。後者はcdnjsには無かったりするが、こういう場合にjsdelivrを使うと大抵なんでも配信してくれるので楽だ。z軸がぎゅっと圧縮されているようになっており、凹凸がわかりづらいので強調させたり、軸の向き、どの平面にグリッドを配置するかを発注者と相談の上調節する。
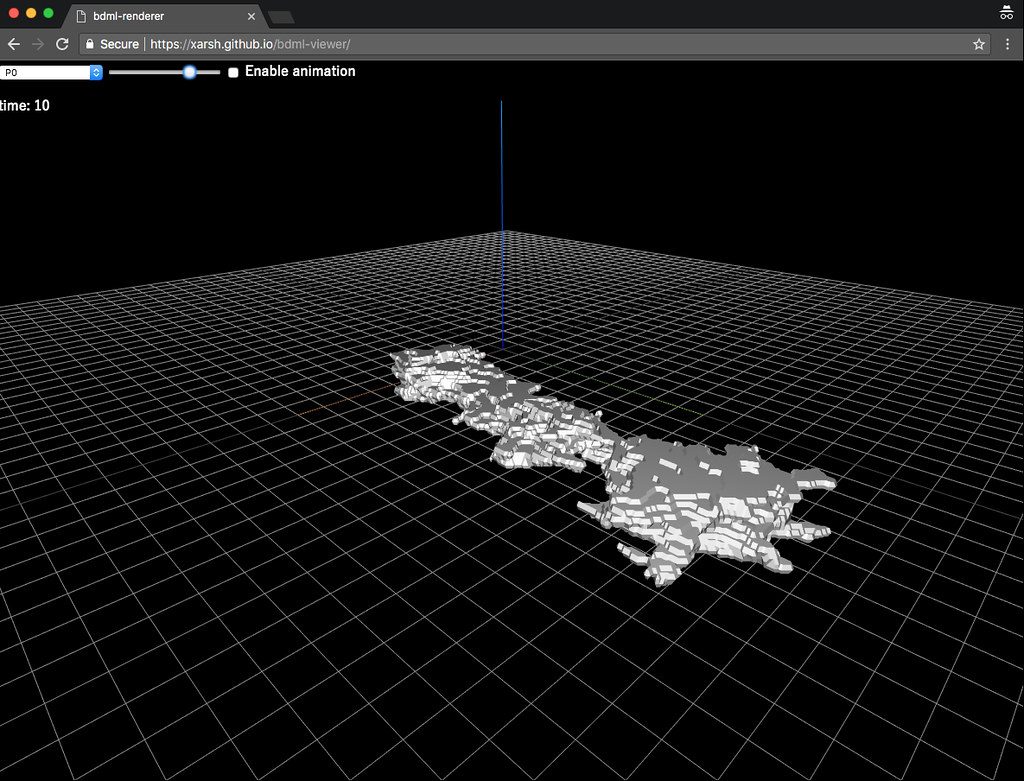
完成したのがこれ。Chromeで動いていることを確認した。
